Apptainer で OpenFold を実行してタンパク質の立体構造予測を試してみた
この記事は公開されてから1年以上経過しています。情報が古い可能性がありますので、ご注意ください。
はじめに
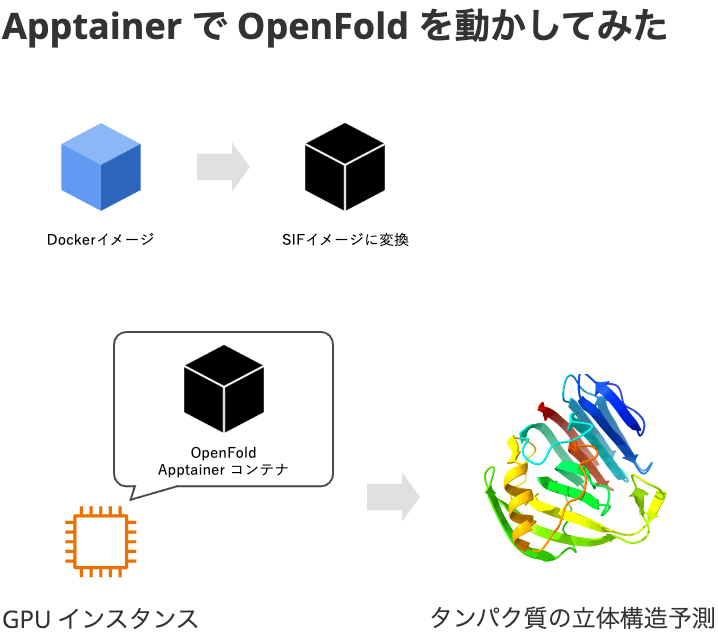
本記事では、Apptainer を使用してタンパク質の立体構造予測できる OpenFold の実行環境の構築方法と注意点を説明します。この分野ではあれば Docker より Apptainer の方が需要ありそうなので試してみました。
Icons made by Freepik from Flaticon
前提知識
OpenFold の基本的な実行方法は以下の記事を参照してください。
検証環境
推論用の GPUインスタンス
OpenFold を実行(推論)するインスタンスは GPU インスタンスを利用します。OpenFold は現在 CUDA 11 と 12 をサポートしています。CUDA 12 の実行環境がセットアップ済みの AMI を利用しました。
以下の環境で検証を実施しました。
- インスタンスタイプ:g6.4xlarge
- NVIDIA L4 Tensor Core GPU を 1 台搭載
- vCPU 16 コア, 64GB メモリ
- AMI:Deep Learning Base OSS Nvidia Driver GPU AMI (Ubuntu 22.04)
- OS:Ubuntu 22.04 LTS
- CUDA Version:12.4
- Apptainer Version: 1.3.6
Apptainer の実行環境構築
最新のインストール手順は公式ドキュメントを参照してください。
Apptainer のインストール
本検証では以下の手順でインストールしました。
sudo apt update
sudo apt install -y wget
cd /tmp
wget https://github.com/apptainer/apptainer/releases/download/v1.3.6/apptainer_1.3.6_amd64.deb
sudo apt install -y ./apptainer_1.3.6_amd64.deb
インストール後、動作確認を行います。
$ apptainer version
1.3.6
$ apptainer run docker://sylabsio/lolcow:latest
INFO: Converting OCI blobs to SIF format
INFO: Starting build...
Copying blob 16ec32c2132b done |
Copying blob 5ca731fc36c2 done |
Copying config fd0daa4d89 done |
Writing manifest to image destination
2024/12/29 22:16:47 info unpack layer: sha256:16ec32c2132b43494832a05f2b02f7a822479f8250c173d0ab27b3de78b2f058
2024/12/29 22:16:47 info unpack layer: sha256:5ca731fc36c28789c5ddc3216563e8bfca2ab3ea10347e07554ebba1c953242e
INFO: Creating SIF file...
______________________________
< Sun Dec 29 22:16:55 UTC 2024 >
------------------------------
\ ^__^
\ (oo)\_______
(__)\ )\/\
||----w |
|| ||
注意: パッケージインストールに失敗
以前、ParallelCluster の Ubuntu 22.04 に Apptainer をインストールしたときはパッケージの追加が可能でした。今回は Deep Learning AMI を使用したせいかこの方法ではインストールできませんでした。
OpenFold コンテナイメージの準備
作成済みの OpenFold の Docker イメージが必要です。OpenFold の Docker イメージの作成方法は以下の記事を参考にしてください。
Apptainer は Docker イメージの実行も可能ですが、今回は Apptainer の用のコンテナイメージ(Singularity Image Format Images)に変換します。
$ docker images
REPOSITORY TAG IMAGE ID CREATED SIZE
openfold latest a79640906979 5 days ago 25.6GB
Docker イメージから Apptainer 用のイメージへの変換
Docker イメージを Apptainer で実行可能な SIF ファイルに変換します。
apptainer buildのあとは、作成するイメージ名の指定です(拡張子は.sifが無難です)docker-daemon:のあとに[リポジトリ名]:[タグ]を指定します
sudo apptainer build openfold.sif docker-daemon://openfold:latest
2024/12/30 01:47:13 info unpack layer: sha256:bd9ddc54bea929a22b334e73e026d4136e5b73f5cc29942896c72e4ece69b13d
2024/12/30 01:47:13 info unpack layer: sha256:b43f70fd2c45daecd1bcc078f594b7db3e2d2c51426c9f05a3ba73c0c763bded
INFO: Creating SIF file...
INFO: Build complete: /mnt/ebs/openfold.sif
13GB のイメージになりました。Docker イメージとは異なり、1 つのファイルとしてコンテナイメージを扱えます。
$ du -h openfold.sif
13G openfold.sif
注意: ディスク容量の確保
変換時には/var配下に十分なディスク容量が必要です。ディスク容量が不足する場合は以下のようなエラーが発生します。私はルートの EBS の容量を 80GB から 100GB に拡張して対応しました。
FATAL: While performing build: conveyor failed to get: while converting reference:
loading image from docker engine: Error response from daemon: write
/var/lib/docker/tmp/docker-export-655311700/blobs/sha256/0dcbb9b968ca4d29228c6a7c1f194fe3226d734b310574bb71ed413165c5e263:
no space left on device
Apptainer で OpenFold コンテナ実行
Docker コンテナで実行するときはdocker runでしたが、Apptainer 様式に置き換えます。
実行コマンド
以下のコマンドで OpenFold を実行します。パスは適時環境に合わせて変更してください。
IMAGE_PATH: Apptaienr 用のイメージファイルのパスを指定してくださいDATABASE_PATH: 必要なデータベースファイルのパスを指定してください- カレントリディレクトリの指定は、Apptainer だと自動マウントされるため不要だったかもしれないです
# 環境変数の設定
IMAGE_PATH="/mnt/s3/images"
DATABASE_PATH="/mnt/ebs/openfold/data"
CURRENT_DIR=$PWD
cd /home/ubuntu/openfold/examples/monomer
mkdir results
# Apptainer 実行コマンド
nohup apptainer exec --nv \
--bind ${CURRENT_DIR}:/local,${DATABASE_PATH}:/database \
${IMAGE_PATH}/openfold.sif \
python3 /opt/openfold/run_pretrained_openfold.py \
/local/fasta_dir \
/database/pdb_mmcif/mmcif_files/ \
--uniref90_database_path /database/uniref90/uniref90.fasta \
--mgnify_database_path /database/mgnify/mgy_clusters_2022_05.fa \
--pdb70_database_path /database/pdb70/pdb70 \
--uniclust30_database_path /database/uniclust30/uniclust30_2018_08/uniclust30_2018_08 \
--bfd_database_path /database/bfd/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt \
--jackhmmer_binary_path /opt/conda/bin/jackhmmer \
--hhblits_binary_path /opt/conda/bin/hhblits \
--hhsearch_binary_path /opt/conda/bin/hhsearch \
--kalign_binary_path /opt/conda/bin/kalign \
--openfold_checkpoint_path /database/openfold_params/finetuning_ptm_2.pt \
--config_preset "model_1_ptm" \
--model_device "cuda:0" \
--output_dir /local/results &
主な変更点
- 実行コマンド:
docker run→apptainer exec - ボリュームマウント:
-v→--bind - GPU オプション:
--gpus all→--nv
実行結果確認
Apptainer でも問題なく実行できました。
- 推論処理: 約 53.6 秒
- リラクゼーション: 約 19.8 秒
[2025-01-03 19:47:18,785] [INFO] [real_accelerator.py:161:get_accelerator] Setting ds_accelerator to cuda (auto detect)
INFO:/opt/openfold/openfold/utils/script_utils.py:Loaded OpenFold parameters at /database/openfold_params/finetuning_ptm_2.pt...
INFO:/opt/openfold/run_pretrained_openfold.py:Generating alignments for 6KWC_1...
INFO:/opt/openfold/openfold/utils/script_utils.py:Running inference for 6KWC_1...
INFO:/opt/openfold/openfold/utils/script_utils.py:Inference time: 53.64408176899997
INFO:/opt/openfold/run_pretrained_openfold.py:Output written to /local/results/predictions/6KWC_1_model_1_ptm_unrelaxed.pdb...
INFO:/opt/openfold/run_pretrained_openfold.py:Running relaxation on /local/results/predictions/6KWC_1_model_1_ptm_unrelaxed.pdb...
WARNING:root:Warning: importing 'simtk.openmm' is deprecated. Import 'openmm' instead.
INFO:/opt/openfold/openfold/utils/script_utils.py:Relaxation time: 19.831181259999994
INFO:/opt/openfold/openfold/utils/script_utils.py:Relaxed output written to /local/results/predictions/6KWC_1_model_1_ptm_relaxed.pdb...
標準出力に吐かれた実行時間のログは、出力結果のディレクトリ内に作成されるtimings.jsonファイルでも確認できます。
$ tree
.
├── alignments
│ └── 6KWC_1
│ ├── bfd_uniclust_hits.a3m
│ ├── hhsearch_output.hhr
│ ├── mgnify_hits.sto
│ └── uniref90_hits.sto
├── predictions
│ ├── 6KWC_1_model_1_ptm_relaxed.pdb
│ ├── 6KWC_1_model_1_ptm_unrelaxed.pdb
│ └── timings.json
└── timings.json
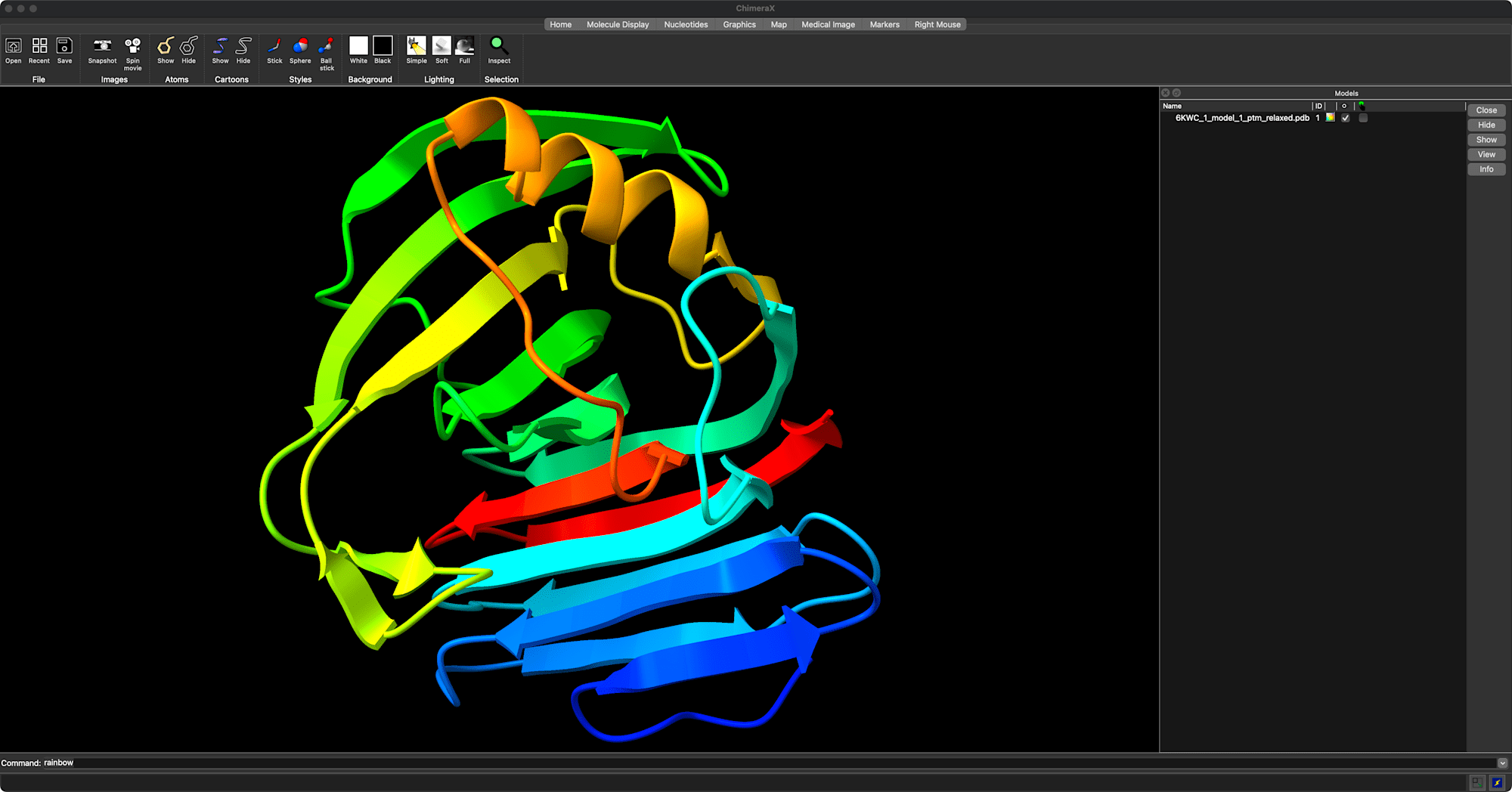
6KWC_1_model_1_ptm_relaxed.pdbを ChimeraX で開いてみました。問題なく読み込めています。
まとめ
- Apptainer でも OpenFold を問題なく実行できました
- Docker と Apptainer の実行コマンドのオプションの違いに注意が必要です
おわりに
コンテナの実行基盤として Apptainer を採用している環境であれば、Apptainer に寄せてしまった方が管理しやすいかもしれません。